Uma nova abordagem em várias camadas descobre 50 novos genes candidatos para a doença de Parkinson

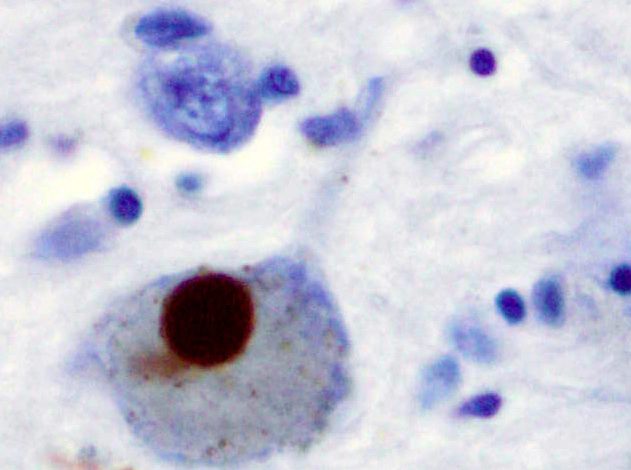
Imuno-histoquímica para alfa-sinucleína mostrando coloração positiva (marrom) de um corpo de Lewy intraneural na substância negra na doença de Parkinson. Crédito: Wikipédia
Muitos distúrbios neurodegenerativos, como a doença de Parkinson (DP), resultam dos efeitos combinados de mutações em vários genes (ou seja, poligênicos). Embora estudos anteriores tenham identificado alguns genes responsáveis por casos familiares ou esporádicos de DP, ainda estamos longe de conhecer todo o espectro de genes que contribuem para esse complexo distúrbio.
Pesquisadores do Instituto de Pesquisa Neurológica Jan e Dan Duncan do Hospital Infantil do Texas e do Baylor College of Medicine desenvolveram recentemente uma abordagem genômica funcional integrada que levou à descoberta de 50 genes que demonstraram pela primeira vez modificar a patologia da DP em um animal doente. modelo. O estudo foi publicado em Genética Molecular Humana.
O estudo foi liderado pelo Dr. Juan Botas, professor do Baylor College e investigador do Duncan NRI. O destaque do estudo é uma nova abordagem multidisciplinar alto rendimento abordagem que a equipe desenvolveu para identificar e validar funcionalmente dezenas de genes neuroprotetores e causadores de DP.
Normalmente, leva vários anos para identificar e validar funcionalmente o papel de um gene em um desordem genética, e esta é uma tarefa particularmente onerosa para uma doença poligênica como a DP. Ao integrar várias abordagens biológicas computacionais e in vivo dentro de uma única estratégia de triagem, a equipe conseguiu identificar e validar muitos candidatos a genes PD em um tempo bastante curto.
Desde 2005, os Genome-Wide Association Studies (GWAS) têm sido usados para analisar os genomas de um grande número de indivíduos para identificar variantes genômicas que estão estatisticamente associadas ao aumento do risco de um distúrbio genético complexo. Embora este método revele loci/variantes genéticas que podem estar potencialmente associadas a uma determinada doença, são necessários estudos mais aprofundados in vitro em células cultivadas e/ou in vivo em modelos animais para demonstrar o envolvimento biológico dessas variantes na patogênese dessa doença, que são processos trabalhosos e demorados.
Nos últimos anos, uma nova abordagem conhecida como estudo de associação ampla do transcriptoma (TWAS) foi desenvolvida para prever o risco genético para doenças complexas. A combinação de TWAS e GWAS com um algoritmo de aprendizado de máquina deu aos pesquisadores insights sobre a função potencial dessas variantes. No entanto, os genes identificados por ambos os métodos precisam de mais validação experimental.
Para acelerar o processo de validação do gene, o principal autor deste estudo, o estudante de pós-graduação Jiayang Li e outros desenvolveram uma abordagem de várias etapas que combinou vários métodos de validação computacional e in vivo.
“Primeiro, nomeamos 160 potenciais genes candidatos PD via GWAS e TWAS, que foram analisados usando outras ferramentas computacionais de última geração e levaram a 80 genes PD de alta confiança”, disse Li. “Em segundo lugar, estabelecemos uma ligação entre esses candidatos e a patologia associada à DP, avaliando se os padrões de expressão desses candidatos foram alterados no transcriptoma cerebral e sanguíneo de pacientes com DP. Finalmente, para avaliar as relações funcionais entre esses candidatos e avaliar quais vias biológicas eles estão envolvidos, realizamos várias análises in silico e in vivo que finalmente resultaram em 50 genes de risco de DP e 14 genes potencialmente neuroprotetores”.
“Nosso sucesso na identificação de tantas novas variantes e a notável congruência nos resultados que obtivemos em cada etapa desta tela apoia isso como um método poderoso para identificar e validar novos genes candidatos à DP”, disse o Dr. Botas. “Além disso, desde que a informação genômica esteja prontamente disponível, essa abordagem pode ser aplicada amplamente a uma ampla gama de distúrbios genéticos complexos e, portanto, prevemos que este estudo tenha um amplo impacto em áreas de doenças muito além da DP”.
Outros envolvidos no estudo foram Bismarck Amoh, Emma McCormick, Akash Tarkunde, Katy Zhu, Alma Perez, Megan Mair, Justin Moore, Joshua Shulman e Ismael Al-Ramahi. Eles eram afiliados ao Baylor College of Medicine e Jan e Dan Duncan Neurological Research Institute no Texas Children’s Hospital.
Jiayang Li et al, Integração do estudo de associação ampla do transcriptoma com ensaios de disfunção neuronal fornece evidências genômicas funcionais para genes da doença de Parkinson, Genética Molecular Humana (2022). DOI: 10.1093/hmg/ddac230
Fornecido pelo Hospital Infantil do Texas
Citação: Uma nova abordagem em várias camadas descobre 50 novos genes candidatos para a doença de Parkinson (2022, 3 de outubro) recuperados em 3 de outubro de 2022 em https://medicalxpress.com/news/2022-10-multilayered-approach-candidate-genes-parkinson.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa particular, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins informativos.
Segue as Notícias da Comunidade PortalEnf e fica atualizado.(clica aqui)






