Adaptando modelos de linguagem para rastrear variantes de vírus
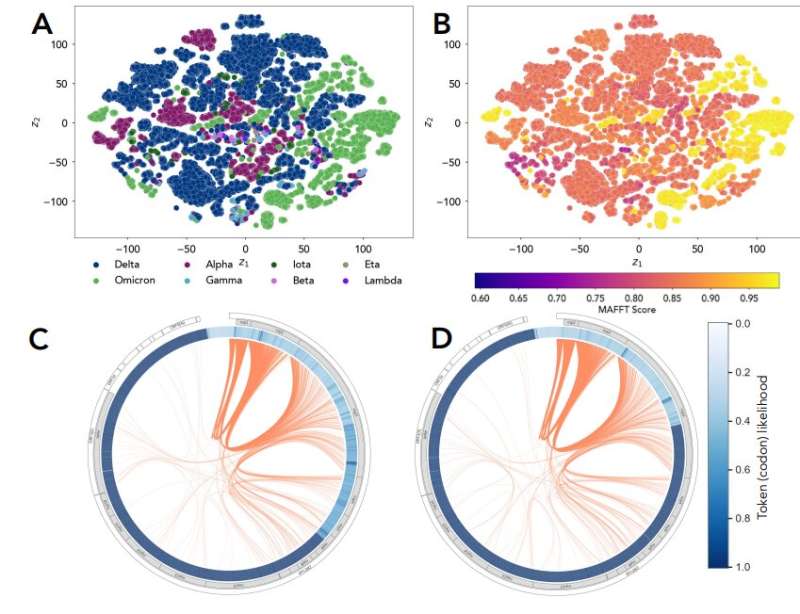
O espaço latente aprendido de GenSLMs descreve propriedades biologicamente significativas para genomas de SARS-CoV-2. (A) As incorporações de GenSLMs são visualizadas com incorporação de vizinhos estocásticos distribuídos t (t-SNE) e cada sequência de gene é representada como um ponto no gráfico 2D. Pintamos cada sequência por seu ID variante – embora tenhamos mais de 515 linhagens PANGO (Rambaut et al., 2020) representadas nos dados, mostramos apenas aquelas com rótulos designados pela OMS. (B) O espaço latente também pode ser pintado com a pontuação de alinhamento determinada por MAFFT (Yamada et al., 2016) em relação a um genoma omicron; o agrupamento nas medidas de distância é claramente visível. A visualização da sequência logarítmica (barra azul) e a atenção cruzada de proteínas (linhas laranja) das cepas (C) Delta e (D) Omicron SARS-CoV-2 destaca como os padrões coevolutivos são diferentes nessas linhagens. É interessante notar que enquanto a proteína Spike da linhagem delta mostra acoplamento com nsp3, nsp5 e outras proteínas, esses acoplamentos não são observados na linhagem omicron. Crédito: bioRxiv (2022). DOI: 10.1101/2022.10.10.511571
Cientistas do Laboratório Nacional Argonne do Departamento de Energia dos EUA (DOE) e uma equipe de colaboradores ganharam o prêmio Prêmio Especial Gordon Bell 2022 para Pesquisa COVID-19 baseada em computação de alto desempenho por seu novo método de identificar rapidamente como um vírus evolui. Seu trabalho no treinamento de grandes modelos de linguagem (LLMs) para descobrir variantes do SARS-CoV-2 tem implicações para a biologia além do COVID-19.
Uma forma de inteligência artificial (IA), os LLMs geralmente são destinados a resumir e traduzir textos ou prever quais palavras podem vir a seguir com base no que o modelo aprendidas numa fase inicial de formação. Por exemplo, um LLM pode ser treinado – com a ajuda de gigantescos conjuntos de dados de idiomas – para traduzir textos do inglês para o espanhol.
Os pesquisadores que ganharam o prêmio deste ano aproveitaram os poderosos recursos de supercomputação e IA da Argonne para desenvolver e aplicar LLMs para rastrear como um vírus pode se transformar em variantes mais perigosas ou mais transmissíveis.
Quando um vírus evolui, ele se transforma em novas variantes que podem ser semelhantes às variantes anteriores ou ainda mais mortais do que as iterações anteriores. Quando uma variante específica é considerada mais perigosa ou prejudicial, ela é rotulada como uma variante preocupante (VOC). Descobrir esses VOCs de forma rápida e eficiente pode salvar vidas, fornecendo aos cientistas tempo para projetar e desenvolver vacinas e estratégias de tratamento eficazes.
Os métodos existentes para rastrear essas variantes podem ser lentos. Para resolver esse problema, o biólogo computacional Arvind Ramanathan e seus colegas da Argonne juntamente com colaboradores da University of Chicago, NVIDIA, Cerebras Inc., University of Illinois at Chicago, Northern Illinois University, California Institute of Technology, New York University and Technical University de Munique decidiu criar um meio de identificar VOCs. o papel deles“GenSLMs: modelos de linguagem em escala genômica revelam a dinâmica evolucionária do SARS-CoV-2”, é o ponto culminante das descobertas da equipe.
“Quando a pandemia começou, tínhamos várias dessas variantes realmente prejudiciais do vírus, como a variante Delta”, disse Ramanathan. “Resultou em um grande número de mortos. Mas o Delta evoluiu como consequência de certas mutações que aconteciam quando o vírus enfrentava os hospedeiros humanos. É um processo de evolução do vírus dentro da célula humana.”
Seu trabalho resultou no primeiro modelo de linguagem em escala genômica (GenSLM), que é um modelo que pode analisar genes e identificar rapidamente VOCs. O modelo discutido no artigo foi treinado com dados da pandemia do COVID-19, e a esperança é que modelos como esse possam fornecer às autoridades de saúde as ferramentas necessárias para responder rapidamente às variantes crescentes. O GenSLM é o primeiro modelo de fundação em escala de genoma que pode ser alterado e aplicado a outras tarefas de previsão semelhantes à identificação de VOC.
Embora essas variantes evolucionárias pareçam surgir aleatoriamente ao olho humano, rastreá-las é a maior preocupação. Dessa forma, o trabalho de Ramanathan e seus colegas pode alterar seriamente a forma como mantemos o controle dos surtos virais.
A linguagem da evolução
Trabalhos anteriores demonstraram que os LLMs baseados na linguagem de aminoácidos das proteínas podem ser usados tanto para rastrear a evolução das proteínas quanto para projetar proteínas inteiramente novas com estrutura e função novas. No entanto, Ramanathan aponta que, até onde sabe, a pesquisa que ele e seus colegas realizaram foi a primeira tentativa de executar um modelo baseado em LLM no nível do gene.
“Modelos de linguagem grandes são essenciais para alcançar a IA para a visão científica em diversos domínios científicos”, disse Venkatram Vishwanath, coautor do estudo e líder de ciência de dados no Argonne Leadership Computing Facility (ALCF), uma instalação do usuário do DOE Office of Science. .
Dito isso, as proteínas ainda estão a dois passos do núcleo processo biológico que Ramanathan e sua equipe estavam interessados. Na célula, os genes são primeiro transcritos em algo chamado RNA mensageiro (mRNA). Esse mRNA sai do núcleo da célula e segue para os ribossomos, onde os ribossomos sintetizam proteínas. De certa forma, você poderia considerar os genes como uma mensagem contendo instruções sobre como construir proteínas. E como as proteínas funcionam de maneira semelhante às mensagens de conversação, faria sentido aplicar modelos de linguagem para defini-las.
Embora experimentos anteriores tenham provado que os modelos de linguagem eram hábeis em explicar as mudanças evolutivas no nível da proteína, os cientistas precisavam ir mais fundo se quisessem identificar VOCs no nível do gene.
O aprendizado de máquina desempenhou um papel fundamental nesta pesquisa, e os modelos precisavam de informações para aprender sobre VOCs. Os recursos da Web do Bacterial and Viral Bioinformatics Resource Center, bem como o Houston Hospital System, forneceram dados integrados e ferramentas de análise para apoiar este trabalho. Os pesquisadores analisaram 1,5 milhão de sequências completas do genoma do SARS-CoV-2 de alta qualidade do centro de recursos e 16.545 sequências totais de Houston para entender melhor o vírus.
Anteriormente, sem esses GenSLMs, os VOCs precisavam ser identificados passando individualmente por cada proteína e mapeando cada mutação para ver se alguma mutação era de interesse. Isso é incrivelmente trabalhoso e demorado, e os GenSLMs devem ajudar a tornar esse processo mais fácil.
A equipe provou que esses modelos podem ajudar no avanço da pesquisa em biologia e agora querem entender até onde podem levar essa abordagem. Ramanathan acredita que seu trabalho pode lançar as bases para um futuro observatório de pandemia. Ele também sugere que as aplicações de engenharia de proteínas podem vir desse trabalho, ou mesmo a modelagem de organismos inteiros.
As ferramentas certas para o trabalho
Poderosos ativos de supercomputação foram vitais para o sucesso deste trabalho. Os pesquisadores usaram o Polaris, um sistema da Hewlett Packard Enterprise, e a plataforma Cerebras CS-2 AI no ALCF. Essa pesquisa também contou com o supercomputador Selene da NVIDIA. Enquanto o Polaris e o Selene são poderosos supercomputadores acelerados por GPUs (unidades de processamento gráfico), o sistema CS-2 é diferente. O sistema acelerador CS-2 AI, parte do ALCF AI Testbed, é altamente otimizado para tarefas baseadas em aprendizado.
“Polaris é o novo supercomputador ALCF com quatro GPUs em um único nó, e temos 560 desses nós”, disse Ramanathan. “Isso realmente nos ajuda a dimensionar o fluxo de trabalho de ponta a ponta, incluindo o processo de treinamento, em vários nós de uma maneira muito mais conveniente. E devido à quantidade de memória e armazenamento local de nó disponível em um único nó, pode carregar ou podemos basicamente organizar os dados de certas maneiras que nos permitem utilizar todo o poder da máquina para fazer esses tipos de cálculos complexos.”
Dada a crise crítica de tempo para obter resultados, a equipe também contou com as máquinas Cerebras CS-2 para auxiliar em seu trabalho, além dos sistemas Polaris e Selene. Especificamente, eles utilizaram o Cerebras Wafer-Scale Engine e acabaram exigindo uma única máquina CS-2, bem como um cluster de 16 CS-2 para alcançar os resultados desejados de precisão e perplexidade em menos de um dia.
“Um desafio chave neste problema é lidar com longos comprimentos de sequência e lidar com esses modelos básicos na escala do genoma viral”, disse Ramanathan. “Esse processo pode se beneficiar de sistemas com grandes recursos de memória, como a arquitetura do sistema CS-2 com sua infraestrutura Memory-X e Swarm-X, e isso facilita o carregamento e o treinamento nessas sequências muito longas”.
“Recursos como Polaris, sistemas CS-2 e os vários sistemas aceleradores de IA no ALCF AI Testbed estão nos ajudando a avançar no uso desses modelos para pesquisa científica”, disse Vishwanath.
Maxim Zvyagin et al, GenSLMs: modelos de linguagem em escala genômica revelam a dinâmica evolucionária do SARS-CoV-2, bioRxiv (2022). DOI: 10.1101/2022.10.10.511571
Fornecido por
Laboratório Nacional de Argonne
Citação: Adaptação de modelos de linguagem para rastrear variantes de vírus (2022, 29 de novembro) recuperado em 29 de novembro de 2022 em https://medicalxpress.com/news/2022-11-language-track-virus-variants.html
Este documento está sujeito a direitos autorais. Além de qualquer negociação justa para fins de estudo ou pesquisa privada, nenhuma parte pode ser reproduzida sem a permissão por escrito. O conteúdo é fornecido apenas para fins informativos.
Segue as Notícias da Comunidade PortalEnf e fica atualizado.(clica aqui)